CodeFasta CodeAA acide_aminé Note_sti_GB Note_inh_GB regexp_inh img_sti img_inh
G Gly Glycine 2A 
E Glu Glutamate 3A 3F CPTV 

K Lys Lysine 3A 3F CPTV 

M Met Méthionine 3A 3F CPTV 

Q Gln Glutamine 3A 3F CPTV 

F Phe Phénylalanine 3B 
U Sel Sélénocystéine 3B 
H His Histidine 3Bb 3E S 

A Ala Alanine 3C 4D W 

S Ser Sérine 3E 3Bb H 

C Cys Cystéine 3F 3A EKMQ 

P Pro Proline 3F 3A EKMQ 

T Thr Thréonine 3F 3A EKMQ 

V Val Valine 3F 3A EKMQ 

D Asp Aspartate 3G 3G DILN 

I Ile Isoleucine 3G 3G DILN 

L Leu Leucine 3G 3G DILN 

N Asn Asparagine 3G 3G DILN 

R Arg Arginine 4C 
Y Tyr Tyrosine 4C 
W Trp Tryptophane 4D 3C A 

nbr nbr nbr nbr nbr nbr nbr
CodeFasta CodeAA acide_aminé Note_sti_GB regexp_inh img_sti img_inh
21.000000 21.000 21.000000 21.000000 16.000000 21.000000 16.000000
4.Tableau proteo image note hominh domaine notes musique jpg
Note_inh_GB regexp_inh note_hominh_img
3f cptv ![]()
3e s ![]()
4d w ![]()
3bb h ![]()
3a ekmq ![]()
3g diln ![]()
3c a ![]()
nbr nbr
Note_inh_GB regexp_inh
7.000000 7.000000
mspaint $BASE\proteodie\fic\jpg\hominh_nb_3f_expreg_cptv. mspaint $BASE\proteodie\fic\jpg\hominh_nb_3e_expreg_s. mspaint $BASE\proteodie\fic\jpg\hominh_nb_4d_expreg_w. mspaint $BASE\proteodie\fic\jpg\hominh_nb_3bb_expreg_h. mspaint $BASE\proteodie\fic\jpg\hominh_nb_3a_expreg_ekmq. mspaint $BASE\proteodie\fic\jpg\hominh_nb_3g_expreg_diln. mspaint $BASE\proteodie\fic\jpg\hominh_nb_3c_expreg_a.
sauvegarde spécifications [proteo_image_note_hominh_tab]
CSV_EDT:copy 5597a7a55408g_dir%\nb_\0.jpg 5597a7a55408g_dir%\hominh_nb_\0_expreg_\1.jpg::copy_image_note_homoinh:
CSV_EDT:hominh_nb_\0_expreg_\1.jpg::nom_image_note_homoinh:
CSV_EDT:mspaint 5597a7a55408g_dir%\hominh_nb_\0_expreg_\1.jpg::edit_image_note_homoinh:
5.Tableau proteo image note
domaine notes musique jpg
Note_sti_GB Note_inh_GB CodeAA_sti regexp_sti regexp_inh note simple sti note simple inh avec nom sti avec nom inh nom acide aminé sti nom acide aminé inh
2A 4F Gly G 





3A 3F Gln EKMQ CPTV 





3B 3Eb Sel FU 





3Bb 3E His H S 





3C 4D Ala A W 





3E 3Bb Ser S H 





3F 3A Val CPTV EKMQ 





3G 3G Asn DILN DILN 





4C 3D Tyr RY 



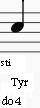

4D 3C Trp W A 





nbr nbr nbr nbr
Note_sti_GB Note_inh_GB regexp_sti regexp_inh
10.000000 10.000000 10.000000 7.000000
filename num basename image
%BASE%/proteodie/fic/jpg/nb_2a.jpg nb_2a 
1 nb_3a 
2 nb_3b 
3 nb_3bb 
4 nb_3c 
5 nb_3d 
6 nb_3e 
7 nb_3eb 
8 nb_3f 
9 nb_3g 
10 nb_4c 
11 nb_4d 
12 nb_4f 
max
filename num
13.000000 12
7.Atelier generation code fasta a partir du code AA
CodeFasta Freq_sti Note_sti_GB
Gly G 220.00 2A
Ala A 261.63 3C Ser S 329.63 3E Val V 349.23 3F Asn N 392.00 3G Gln Q 440.00 3A His H 466.16 3Bb Sel U 493.88 3B Tyr Y 523.26 4C Trp W 587.00 4D nbr
CodeAA
10.000